聚类(clustering)算法是一种十分常用,方便的方法。
首先它是非监督学习,其训练样本没有标记信息。在日常任务中,我们往往会遇到手上有了数据但不知道具体该怎么做的情况,比如一些商业应用中需要对用户的类型进行判别,但具体怎么定义“用户类型”却十分困难。这个时候就需要用到聚类算法,对数据进行聚类之后再根据每一类的特点定义“用户类型”就会简单很多。
聚类算法既能作为一个单独的过程,用于寻找数据内在的分布结构,也可以作为分类等其他学习任务的前驱过程。
1. 度量
在具体讲聚类的算法之前,先让我们来了解一下聚类算法的基础知识,其中最为重要的就是如何进行度量。
在聚类算法中度量可以分为两种——性能度量和距离度量。
1.1 距离度量
距离度量是聚类算法的基础。两个点是否为同一个类,就要看这两个点离的是否“足够近”。
最为常用的距离度量是闵可夫斯基距离(Minkowski distance):
$dist_{mk}(x_i,x_j)=(\sum_{u=1}^{n}|x_{iu}-x_{ju}|^p)^{1/p}$
当P=2时,即为欧式距离(Euclidean distance):
$dist_{ed}(x_i,x_j)=\sqrt{\sum_{u=1}^n|x_{iu}-x_{ju}|^2}$
当P=1时,即为曼哈顿距离(Manhattan distance):
$dist_{man}(x_i,x_j)=\sum_{u=1}^n|x_{iu}-x_{ju}|$
……是不是觉得很熟悉~
另外还有一种针对离散变量的常用距离计算VDM(Value Difference Metric):
$VDM_P(a,b)=\sum_{i=1}^k|\frac{m_{u,a,i}}{m_u,a}-\frac{m_{u,b,i}}{m_u,b}|^P$
其中m{u,a}表示属性u上取值为a的样本数,m{u,a,i}表示在第i个样本簇中在属性u上取值为a的样本数,k为样本簇数。
这里说一下关于变量类型的问题,这个问题在进行属性数值运算时经常遇到。
● 对于连续变量,一般我们会做一下标准化或者归一化。
● 对于离散变量中的有序变量,如{大一,大二,大三}、{满意,一般,不满意}等,我们可以用类似{1,2,3}这样的值去替代,因为这样可以保留更多的信息。可借助sklearn.preprocessing.OrdinalEncoder()来帮助替换。
● 对于离散变量中的无序变量,如{飞机,火车,轮船},{医生,记者,学生}等,我们以往使用onehot编码来替换,但是在计算距离时onehot编码会损失信息,因为各个编码之间的距离是一样的。这时候我们需要使用VDM这类计算方式,来尽量多的保留变量信息。但是有一个问题:大部分的算法库并没有考虑无序变量,这样一来如果我们要实现VDM这类度量,只能自己手动撸了。这使得在要求不高的情况下onehot凑合着用就行了,反正聚类本来就是个大概。(当然如K-prototypes可以处理混合变量,十分周到~)
1.2 性能度量
对数据进行聚类,我们的目标很简单:“簇内相似度(intra-cluster similarity)”高且“簇间相似度(inter-cluster similarity)低”。对一个聚类的结果一般从这两个方面去度量。
而性能度量根据有没有“参考模型”可以分为两种外部指标和内部指标。
1.2.1 内部指标
内部指标就是单纯靠聚类后类内和类间的距离来评估。
常见的有:
● Silhouette Coefficient
Silhouette Coefficient 对每个样本都进行了评估:
$S=\frac{b-a}{max(a,b)} $
a:在同一个类中,一个样本与其他所有点的平均距离。
b:一个样本与下一个最近的类中的所有其他点的平均距离。
最后对数据集的整体评估为每个样本评估的平均值。
1 | ## 优点:取值[-1,1]。负数表示错误分类,正数表示正确分类,0表示重叠分类。 |
● Calinski-Harabaz Index
也被称为Variance Ratio Criterion。
$s(k)=\frac{Tr(B_k)}{Tr(W_k)}*\frac{N-k}{k-1}$
$W_k=\sum_{q=1}^k\sum_{x\in C_q}(x-c_q)(x-c_q)^T$
$B_k=\sum_qn_q(c_q-c)(c_q-c)^T$
其中N为数据的样本数,k为簇数,$C_q$为簇q的样本集,$c_q$为簇q的中心点,c为整个数据的中心点,$n_q$为簇q的样本数。
1 | ## 优点:得分越高表示簇越稠密,分类效果越好 |
● Davies-Bouldin Index
这个指标根据各个簇内距离和簇之间的距离远近来评估。
$R_{i,j}=\frac{s_i+s_j}{d_{i,j}}$
$DB=\frac{1}{k}\sum_{i=1}^kmax_{i\ne j}R_{i,j}$
$s_i$表示簇i中所有的点到簇中心点的平均距离。
$d_{i,j}$表示簇i和簇j的中心点距离。
1 | ## 优势:得分越低越好,最低为0 |
1.2.2 外部指标
外部指标的计算需要依靠“参考模型”,比如领域专家给出的划分结果,所以并不是很常用。
一般这种指标也就看看自己的聚类和别人的聚类是否相似,比如某个领域已经有专家人工对数据进行了分类,现在你用机器学习方法也聚了一下类,然后想看看两者是否相似。
常见的有:
● Adjusted Rand index
该指标计算了预测标签和标记标签的相似度。
首先是一般的Rand index
$RI=\frac{a+b}{C_n^2}$
a:表示在预测中属于同一簇且在参考标记中也属于同一簇的样本对的数量。
b:表示在预测中不属于同一簇且在参考标记中也不属于同一簇的样本对的数量。
$C_n^2$: 表示样本对总数。
但是RI系数有个缺点:当标签是随机分配时,指标并不接近零,这是反直觉的。
因此ARI:
$ARI=\frac{RI-E[RI]}{max(RI)-E[RI]}$
其中,E[RI]为随机标签时,RI的期望。
1 | ## 优势:得分[-1,1]。随机标签得分接近0,负数则效果差,正数则效果好。 |
● Mutual Information
基于互信息,也就是利用信息熵来评估两者的相似程度。
主要分为两种:Normalized Mutual Information(NMI) 和 Adjusted Mutual Information(AMI)
一般AMI比NMI稍好一点。
数学公式有点多,这里就不展开了,实际上本质并没有区别,无非就是用信息熵代替了原来的比值。
1 | ## 优势:得分接近0表示是随机标签。最高为1。正好负坏。 |
● V-measure
使用条件熵分析。
分为三个指标:
homogeneity_score:同质性指标,指每个簇中的样本都来源于一个类。(可以理解为标记分类含括了预测分类)。
completeness_score:完整性指标,指每个类中的样本都来源于一个簇。(可以理解为预测分类含括了标记分类)。
V-measure: 为两者的调和平均数。和上面的NMI基本一个东西~
具体可以看论文:V-Measure: A conditional entropy-based external cluster evaluation measure
1 | ## 优势:[0,1]。0最坏,1最好 |
● Fowlkes-Mallows index
既然是分类问题,为什么不用混淆矩阵来评估呢,都有了标注那和监督型分类的评估也没什么不一样了。
$FMI=\frac{TP}{\sqrt{(TP+FP)(TP+FN)}}$
TP:真阳性。FP:假阳性。FN:假阴性。
1 | ## 优势:随机标签得分接近0.优秀分类得分趋于1. |
2. 算法
聚类算法是一类十分“灵活”,“活跃”的算法。不同的距离度量,不同的评估标准等都会诞生新的聚类算法,现在已知的聚类算法都有100多种了。
当然我们只要最常用,效果最好的几种方法就够了。
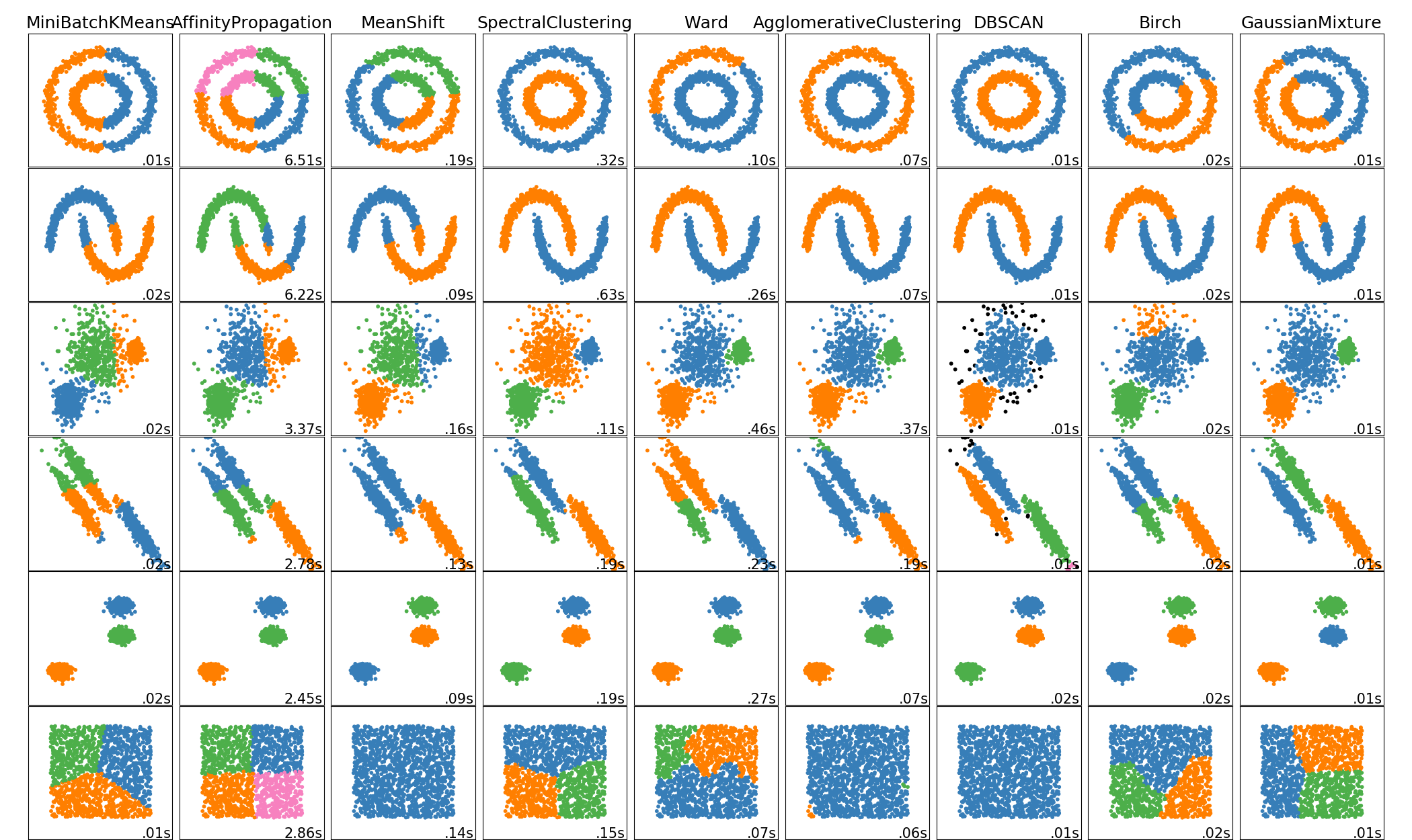
2.1 K-means
k均值可以说是最为流行的聚类算法了。因为他简单、高效、易实现。
k-means算法的思想很简单:将数据分为k类,并使簇内样本相似度最高,即最小化平方误差。
$E=\sum_{i=1}^k\sum_{x\in C_i}||x-u_i||_2^2$
$u_i$为簇i的中心、均值向量。
k-means的目标很明确,但最小化E却是一个NP难问题。 只能通过启发式的,贪心策略来获得近似解。
接下来让我们看一下它是怎么执行的:
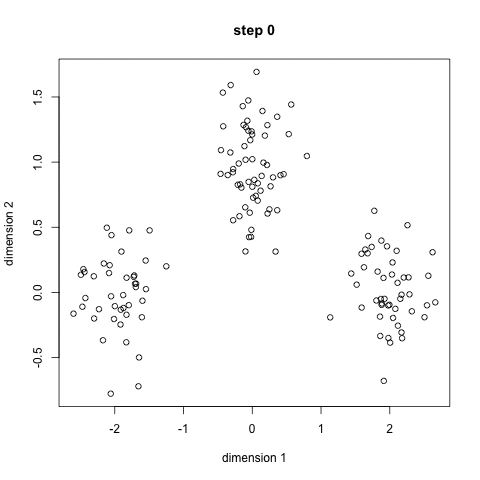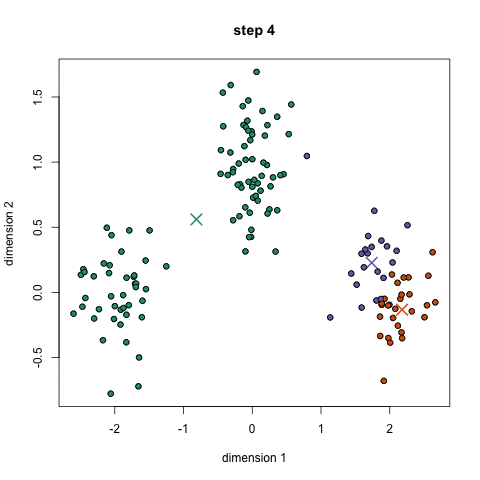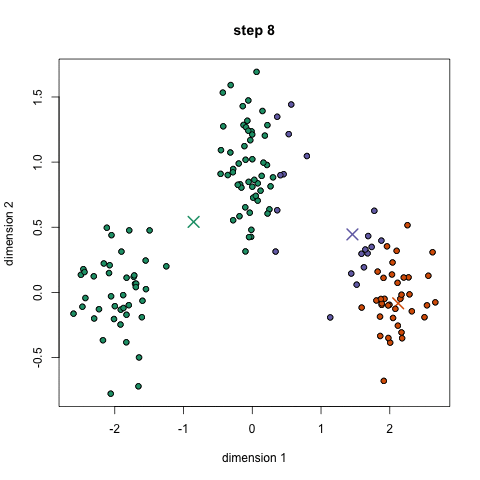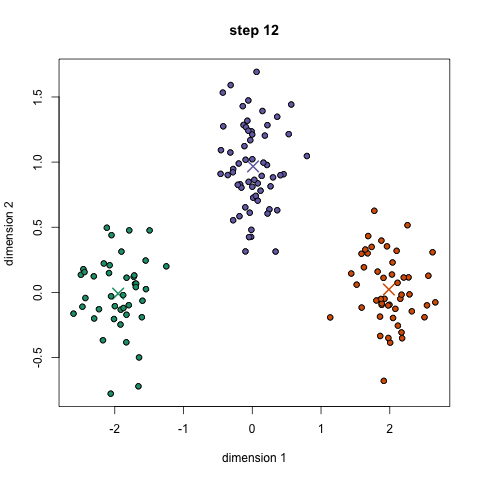
- 首先我们要确定将数据分为几个类,即确定K值。然后随机选取K个点作为簇的中心点。
- 通过计算每个样本与中心点的距离对数据进行分类。
- 根据分类后的数据,可以重新计算每个簇的中心点。
- 不断重复2、3步骤,获得优秀的分类结果。
K-means 的优点
K-means的优点很明显:简单,快速。
K-means的时间复杂度为O(nkt),n是样本数,k是聚类数,t是迭代次数。
K-means 的缺点
● 需要事先确定分几个类。一般只能选取足够大的分类数,导致一些自然的聚类被多个聚类表示。
● 对噪声点比较敏感。可以用LOF算法提前去除离群点,也可以改用k-Mediods算法,用中位数代替均值。
● 需要随机初始化中心点。原始中心点的选取对最终结果的影响十分大。我们需要进行多次不同的初始化来逼近最优解(n_init参数)。sklearn实现了一种称为K-means++的初始化方法,其可以一定程度上解决初始化的问题,它的策略是使初始化的中心点尽量互相远离。
● 相比于其他算法,K-means的结果可复现性差。
● 由于K-means使用欧几里得距离作为距离度量并用均值法来求取中心点,直观的理解,可以认为其在用“圆”来划分数据。也就意味着K-means对于“长条”型的数据分类效果会比较差。
● 由于K-means使用欧几里得距离作为距离度量,这会导致当数据比较高维时度量会膨胀也就是“维度诅咒”。可以在数据预处理时使用PCA技术进行降维,来减轻高维度带来的问题且能提升算法速度。
1 | ## 在sklearn中还实现了MiniBatchKMwans算法,这个算法在一般K-means算法基础上使用了批处理方式,从而不需要## 每次都处理所有数据,这能够大大提升算法的速度,以牺牲一点点分类质量为代价。 |
2.2 Mean-Shift
均值漂移算法是基于滑动窗口的算法。这个算法的目标在于寻找数据的密集区域, 其认为在一个簇的中心点数据一定是最密集的!
让我们看一下它具体是怎么运行的:
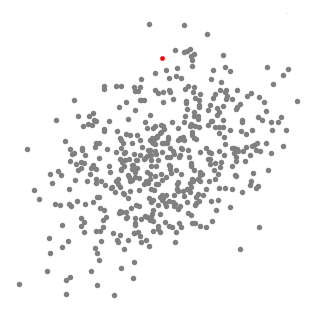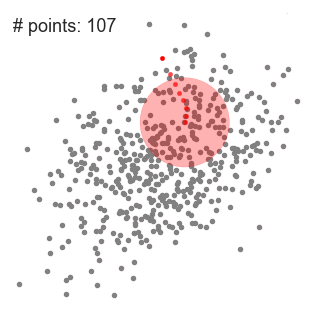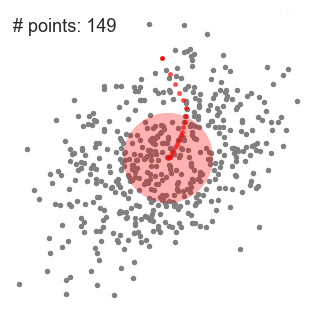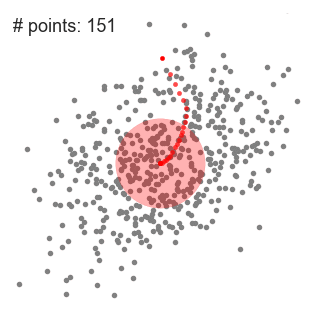

- 首先初始化许多窗口中心点,并设置一个合适的窗口大小。
- 将一个窗口中所有点的均值作为这个窗口新的中心点,这样一来窗口就会向着高密度区域滑动。
- 不断的重复2,直到窗口不再移动为止。
- 将距离相近的中心点进行合并,得到最终分类结果。
mean-shift优点:
● 对噪声有很强的鲁棒性
● 只有一个参数调参方便
mean-shift缺点:
● 窗口宽度大小的选取对最终结果有比较大影响。
● 速度慢,O(n^2)
● 可能会产生一些不确定的小聚类点。
mean-shift的窗口:
有窗口那么就一定会有核函数选择的问题。实际上就是一种花哨的权重分配方法~。而在核函数选择问题中,往往都是Gaussian kernel获胜。但在sklearn中却只实现了Flat kernel,很难受。。

1 | from sklearn.cluster import MeanShift |
2.3 DBSCAN
Density-Based Spatial Clustering of Applications with Noise(DBSCAN)是一种著名的基于密度聚类的算法。
DBSCAN和Mean-Shift比较相似。Mean-Shift认为在一个簇的中心点数据一定是最密集的, 而DBSCAN则认为在两个簇之间数据一定是稀疏的!
那么DBSCAN的重点在于怎么判断一个区域数据是否稀疏。
$minPts:一个区域中最少的数据点数。$
$\epsilon:区域半径。$
可以认为一个点如果在其半径为ε的区域内,数据点的总数少于minPts,那么这个点就是孤立点。
为了更直观的理解,让我们看一下其具体流程:
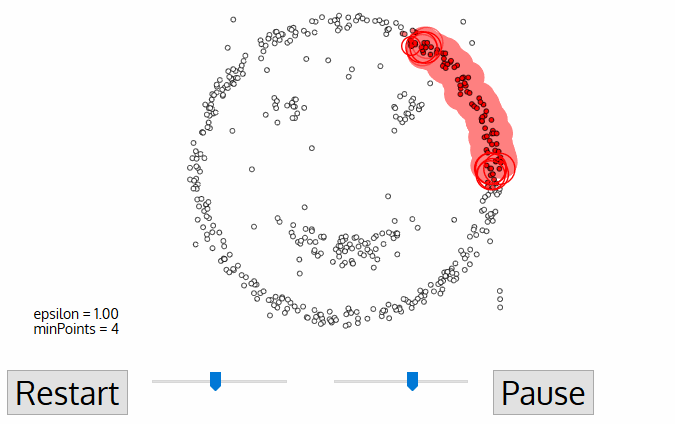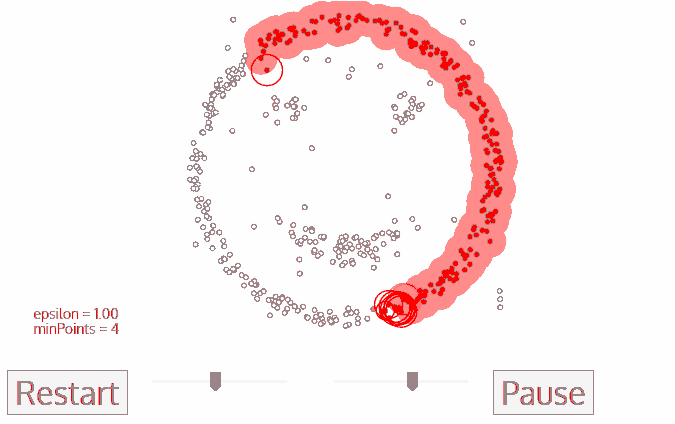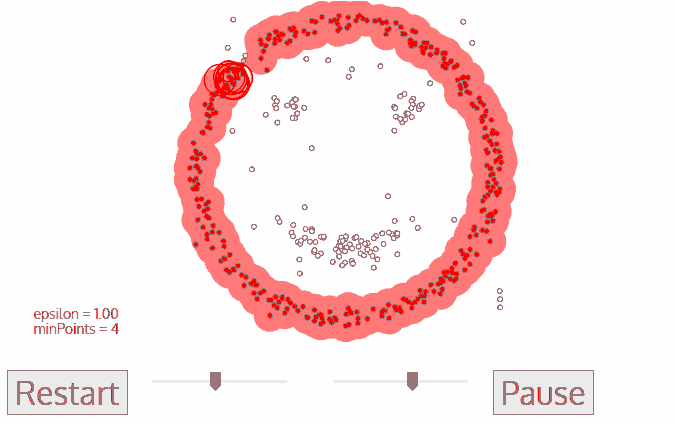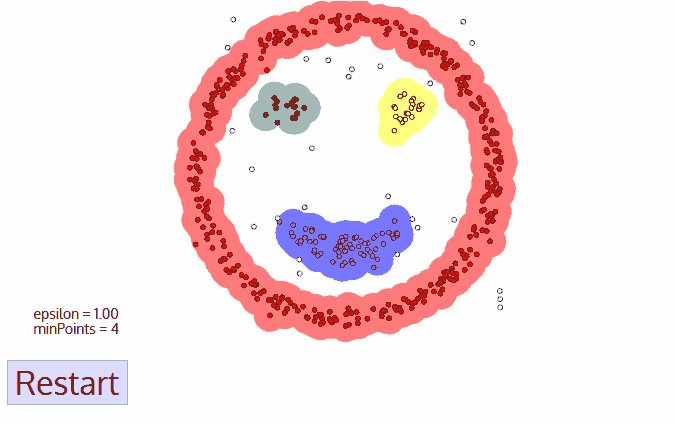
- 遍历所有点,判断其是否为核心对象 。核心对象指的是该点满足:以该点为圆心,ε为半径的区域内数据点数不小于minPts。
- 以任一“未用”核心对象为起点建立一个新簇,其ε半径内的点都为该簇内点。
- 选取该簇内一个“未扩展”的核心对象,其ε半径内的点都为该簇内点。
- 不断重复3,使得簇不断生长,直到簇内的核心对象都已经“扩展”过,即生成一个完整的簇。在执行步骤2。
- 对于那些不在任何簇内的点标记为噪声点。
参数的选择
minPts:
● 一般大于3,大于(维度+1),且一般大一点好。
● 数据集越大,minPts越大。
ε
● 可以通过K-distance graph 来辅助选择。
● 取值太小会导致大量数据无法分类;太大则会导致类别数太少。
● 一般来说,取值小一点较好。
DBSCAN优点:
● 不需要指定簇数。
● 可以对任意形状的稠密数据集进行聚类。
● 能够标记噪声点,对异常点不敏感。
● 只要参数不变,能够重现分类结果。
DBSCAN缺点:
● 当样本集的密度不均匀、聚类间距差相差很大时,聚类质量较差。
● 对于高维数据处理效果较差。
● 速度慢,O(N^2),可以对搜索最近邻时建立的KD树或者球树进行规模限制来改进。
● 两个参数调参很麻烦,不同的参数对最终结果影响很大。
1 | from sklearn.cluster import DBSCAN |
2.4 GMM
Gaussian Mixture Model,GMM, 高斯混合模型 。其认为数据分布符合高斯分布,于是算法就用多个高斯模型去表示、拟合数据的分布!
GMM和K-means十分的像。实际上K-means可以看作是GMM的一种特殊情况。
直观的来看:
● 在K-means中,我们用欧几里得求距离用均值求中心点,实际上就是在用“圆”去分割数据。而GMM则更一般化,其用高斯函数去拟合,高斯函数拥有均值和方差两个变量,可以看成是在用“椭圆”去分割数据。很明显GMM的拟合能力强于K-means。
● 在K-means中,我们将界限划分的很强硬,一个点一定且只能属于一个簇。而GMM则更一般化,因为高斯函数有个好处,其可以用概率大小来表示距离的远近,所以我们可以得到一个点对于各个簇的概率。这对于那些“中间点”的分析友好了太多。
● 求解时,在K-means中,我们使用贪心的策略启发式的去寻找中心点。在GMM也一样,不过因为用的是概率所以称为EM(最大期望)算法,本质上并没有什么区别。
GMM算法主要的变量有4个:
$K$:和K-means算法一样,需要预先确定有几个簇。在GMM也就是component的数量。
$\mu_i$: 第i个高斯函数的均值。
$\Sigma_i$: 第i个高斯函数的协方差。
$\pi_i$: 第i个高斯函数的权重,可估计为$N_k/N$。
其中只有K值需要人为确定,另外的值则通过学习自动获取。
具体流程:
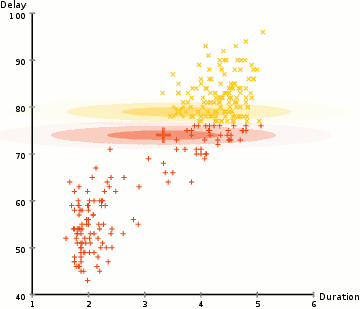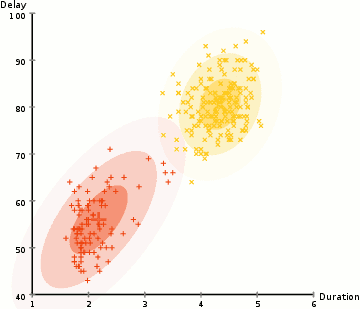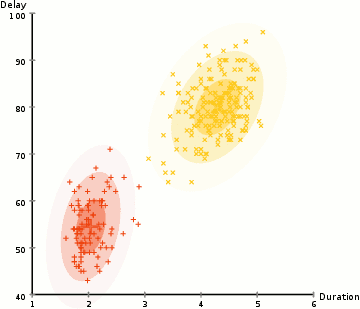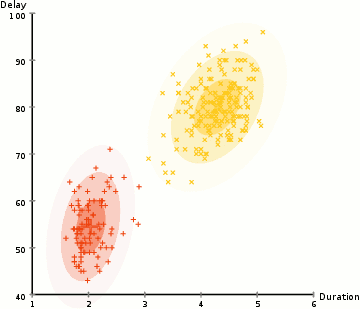
- 首先确定簇的个数K,并初始化高斯分布的参数和权重。
- 根据现有参数计算每个样本属于每个component的后验概率。E步
- 根据极大似然估计(即使得数据整体的概率最大化),更新参数和权重。M步
- 不断重复23步,得到近似最优解。
这里实际上跟着公式来看更加清晰易懂,但是实在懒得打公式了~~
GMM优势:
● 数据点的分类用概率的方式进行描述,对“中间点”更友好。
● 相比于K-means,更灵活,分类效果更好。
● 速度快,O(n)
GMM劣势:
● 需要预先确定K值。
● 需要初始化高斯参数,不同的初始化会对结果产生很大的影响。
1 | from sklearn import mixture |
2.5 Hierarchical
Hierarchical clustering层次聚类算法,也是一种十分常用的算法。
Hierarchical Clustering主要分为两大类:bottom-up、agglomerative、自下而上和 top-down、divisive、自上而下。其中主要使用自下而上的算法,因为自上而下算法执行起来太麻烦。。
自下而上的算法又被称为Hierarchical Agglomerative Clustering,HAC,凝聚式层次聚类。其首先将每个数据点视为一个单一的簇,然后连续地合并(或聚合)两个簇,直到所有的簇都合并成一个包含所有数据点的簇。
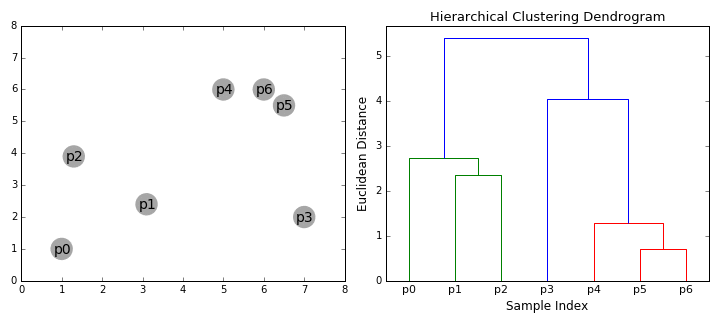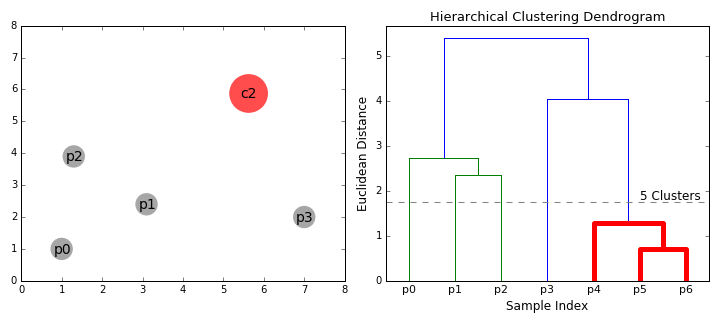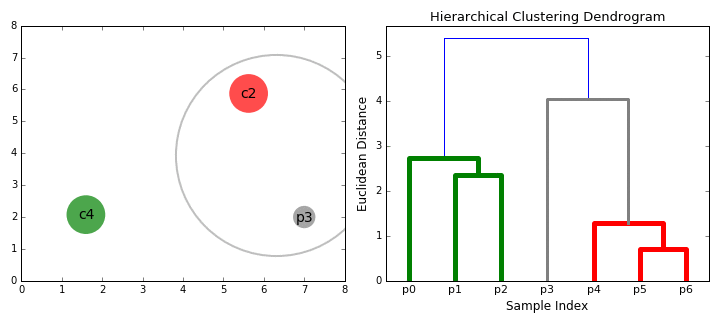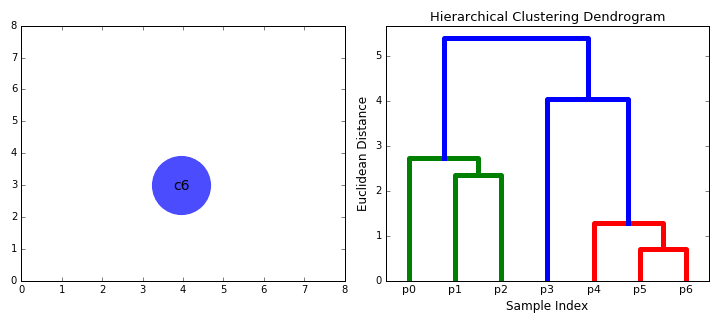
那么整个算法的主要问题就是:怎么计算两个点的距离和怎么选择簇进行合并。
如何计算两个点的距离
在前面距离度量中已经提过这个问题,一般选用欧式距离(“euclidean”,“l2”)。
也可以选用其他度量方式,如:manhattan(“Cityblock”,”l1”),cosine,precomputed。
怎么选择簇进行合并
可以将距离最近的两个类进行合并:
● Single linkage: 两个类中距离最近的点的距离。
● Maximum or complete linkage: 两个类中距离最远的点的距离。
● Average linkage: 将两个类中所有点的距离进行平均。
也可以选择合并后指标最小的情况:
● ward: 和K-means的思想相似,最小化簇内的平方误差。
sklearn中对这些合并策略、指标进行了分析:
● ward的效果最好,但是其距离度量无法改变,只能用欧氏距离。
● 在非欧式距离情况下,Average linkage表现最好。
● 在非欧式距离度量中:“l1“距离对稀疏特征和噪声往往有比较好的效果。
● 可以进行连通性约束,即只有“相邻”的簇可以合并。
Agglomerative Hierarchical的优点:
● 不需要指定簇的数量,甚至可以选择看起来较好的分类。
● 对于距离度量标准的选择并不敏感,效果都比较好。
缺点:
● 其优秀的聚类能力以效率为代价,O(n^3)。
1 | from sklearn.cluster import AgglomerativeClustering |
2.6 Spectral Clustering
因为谱聚类属于用起来很简单,但证明起来有点麻烦的算法,涉及的东西有点多,所以这里不讲具体的证明,只讲一些简单的概念和使用。具体的证明可以看刘建平的博客或者谱聚类的论文 。实际上整体看下来并不难,只是会感觉真的精彩。
谱聚类(spectral clustering)是广泛使用的聚类算法,相比于K-means,其速度更快,效果更好!
谱聚类,名字很高大上,但其实思想并不难。其重点在于对数据无向图的切割,这点和DBSCAN有点像。我们可以将数据用无向图的形式表现出来,顶点表示样本点,边表示数据之间的权重。当我们要将数据分成K份时,实际上就是将该图切割为K个不相连的字图,我们的策略就是尽可能的使被切断的边的权重最小。 而图往往用矩阵来表示,然后经过一系列十分神奇的转换后,这个问题可以用很简单的矩阵运算求得近似最优解!
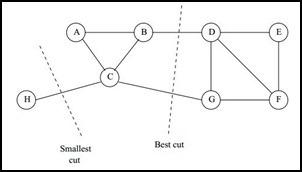
接下来,简单讲一下一些基本概念:
怎么得到权重?
权重的获得分为两部分:哪些点之间的权重需要表示?权重怎么用具体数值表示?
边的选取:
● ε-临近法:对距离为ε内的点进行权重计算。
● K-临近法:对距离最近的K个点进行权重计算。
● 全连接:对所有的点进行权重计算。
其中,无疑全连接的效果是最好的,用的也最普遍。
权重表示:
● 多项式核函数
● 高斯核函数
● Sigmoid核函数
就是将距离进行变换来表示权重,其中高斯核RBF最常使用。
怎么用矩阵表示图
权重矩阵、邻接矩阵W: 矩阵中的每个值$\omega_{i,j}=\omega_{j,i}$表示点i和j的权重。
度矩阵D:对角矩阵,只有主对角线有值。值为该点到其他所有点的权重的和。
拉普拉斯矩阵L: L=D-W。其特性有助于推导证明。
如何切割无向图
切割的关键点在于使得被切割的权重最小。
● 直接将被切割的权重相加。但是容易切割出孤立点。
● RatioCut:在权重相加的基础上除以该子集的点个数,即考虑最大化每个字图的点数。
● Ncut:在权重相加的基础上除以该子集的权重和。
RatioCut和Ncut实际上都考虑了对字图“大小”的度量,防止孤立点的出现。其中Ncut更常用一些。
如何获得最优切割方案
已经有了最小化的目标:RatioCut或Ncut。但很不幸这是个NP难问题,无法直接求解。于是在这里谱聚类用了一个十分巧妙的转换,利用拉普拉斯矩阵的特性将这个问题变为求解k个最小特征值的问题!
流程
- 根据数据构造一个 Graph ,Graph 的每一个节点对应一个数据点,将相似的点连接起来,并且边的权重用于表示数据之间的相似度。把这个 Graph 用邻接矩阵的形式表示出来,记为 W。
- 把W的每一列元素加起来得到
 个数,把它们放在对角线上(其他地方都是零),组成一个N*N的矩阵,记为 D。并令 L=D-W 。
个数,把它们放在对角线上(其他地方都是零),组成一个N*N的矩阵,记为 D。并令 L=D-W 。 - 求出L最小的K个特征值以及对应的特征向量 。
- 把这k个特征(列)向量排列在一起组成一个N*K的矩阵,将其中每一行看作K维空间中的一个向量,并使用 K-means算法进行聚类。聚类的结果中每一行所属的类别就是原来数据点的分类结果。
整体
想要更好的理解整个算法必须跟着公式一步步证明下来。但如果换个角度,结合降维算法可以将其看成对数据进行Laplacian Eigenmap降维之后再做一个传统聚类,而这个传统聚类一般选择K-means! 我们知道,传统的聚类算法对于高维数据的处理结果往往不好,而谱聚类简单来说就是先进行了降维再处理,于是性能得到了情理之中的提升!
1 | from sklearn.cluster import SpectralClustering |
3. 后记
在这里记录一些陆续看到的、感觉挺有启发的tips。
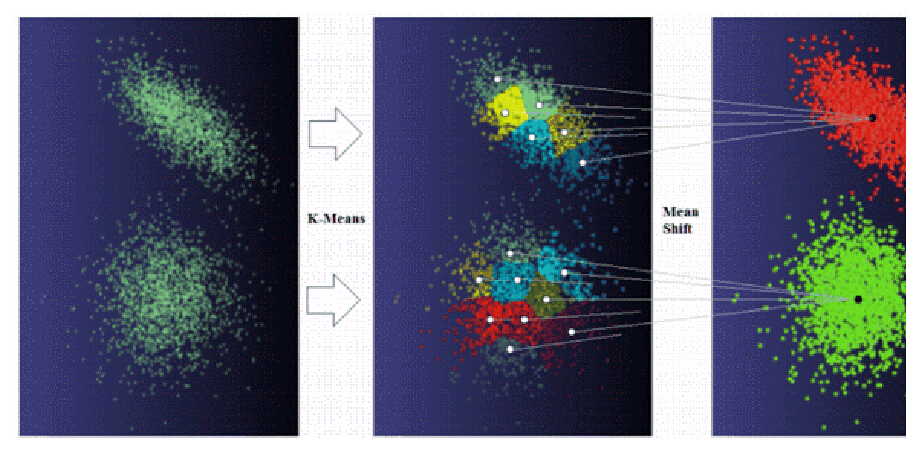
3.1 K-means + Mean-shift
我们知道K-means有个很大的问题就是K值难以确定,而Mean-shift的问题就是速度慢,会产生一些小的不确定聚类点。那么将两者进行结合就能克服这些问题。
首先用K-means进行聚类,结果可能出现一个自然类被分成了好几个小类的情况,这时将聚类点作为Mean-shift的窗口中心点,这样就会将一些合适的聚类点进行合并,解决了K值难以确定的问题; 另外由于由K-means确定了mean-shift的初始窗口中心,使得计算量可以大大的下降;另外由于K-means得到的聚类点一般位于高密度空间,解决了出现小的不确定聚类点的问题;另外两者的结合还能解决非凸聚类的寻找问题。
4. 参考资料
● https://towardsdatascience.com/the-5-clustering-algorithms-data-scientists-need-to-know-a36d136ef68
● https://scikit-learn.org/stable/modules/clustering.html
● https://www.slideshare.net/MahbuburShimul/dbscan-algorithom
● http://blog.pluskid.org/?page_id=78
● https://www.cnblogs.com/pinard/p/6221564.html
● https://www.cs.cmu.edu/~aarti/Class/10701/readings/Luxburg06_TR.pdf
● Li, James (2012). “Visualizing High Dimensional Data”